| 时间:2024-01-19 |
2021年发表于《Molecular Therapy-Nucleic Acids》(IF =8.8)
一、研究背景
青春期是成长发育的重要时期,神经内分泌系统会发生一系列复杂、综合的变化。然而,弓形核(ARC)在青春期开始时转录重编程的调控机制仍不清晰。为了了解表观遗传在调节基因表达中的作用,分离小鼠4周和8周时下丘脑弓形核(ARC),通过RNA-seq、简化甲基化测序RRBS-seq、简化羟甲基化谱RRHP-seq和染色质可及性测序ATAC-seq,发现逆转录因子(REs)的总体DNA甲基化和DNA羟甲基化变化与青春期ARC中的基因表达模型相关。本研究重点分析了5-羟色胺受体6 (Htr6)基因启动子和KISS-1转移抑制基因增强子上的两个短穿插核元件(SINEs)的DNA羟甲基化和DNA甲基化,并研究了它们在基因表达中的调控作用。本研究揭示了一种新的表观遗传机制,SINEs通过这种机制调节青春期的基因表达。
二、研究结果
1、青春期ARC中DNA羟甲基化和REs甲基化图谱
研究了5-甲基胞嘧啶(5mC)和5-羟甲基胞嘧啶(5hmC)在蛋白质编码基因启动子上的富集变化。4周龄和8周龄ARC之间5mC水平显著不同的REs(图1A)。30.4%的差异甲基化区(DMRs)分布在重复元件中,包括584个LINEs、93个低复杂度序列、408个LTRs、4个satellites、200个简单重复序列和522个SINEs,分布在4- 8周龄的ARC之间。同样,51.8%的差异羟甲基化区(DHMRs)包含22555个LINEs,7539个低复杂度序列,24233个LTRs, 360个satellites,17,872个简单重复序列和46,044个SINEs,如图1B所示。此外,在4周龄和8周龄ARC中年DNA甲基化和羟甲基化的变化在REs中具有完全相反的趋势。这些结果表明,基因组REs在青春期的DNA甲基化和羟甲基化,可能参与青春期基因表达模式的调控。
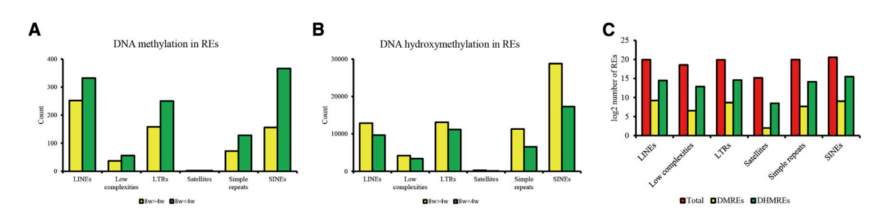
图1 青春期ARC中逆转录元件(RE)的全基因组DNA羟甲基化和DNA甲基化
2、编码区REs的DNA甲基化在调控基因表达中的作用
进一步分析了DNA羟甲基化差异位点(DHMREs)和DNA甲基化差异位点(DMREs)的位置,发现46.0%的DMREs和51.4%的DHMREs位于DEGs附近或内部,这些位点与突触连接、神经元发育、激素反应和表观遗传调控密切相关(图2A和2B)。REs启动子的DNA甲基化与基因表达呈强正相关,而外显子REs的DNA甲基化与RNA水平之间没有统计上的的相关性(p < 0.05)。 然而5'UTR和“下游元件”(3'UTR下游3 kb以内)太少,相关系数无法统计确定(图2C)。因此,REs的DNA羟甲基化与基因表达不相关(图S1)。基因启动子中REs的DNA甲基化与基因表达之间的正相关可能与传统结果相矛盾,传统结果是相邻基因的转录通常被SINEs的DNA甲基化抑制。
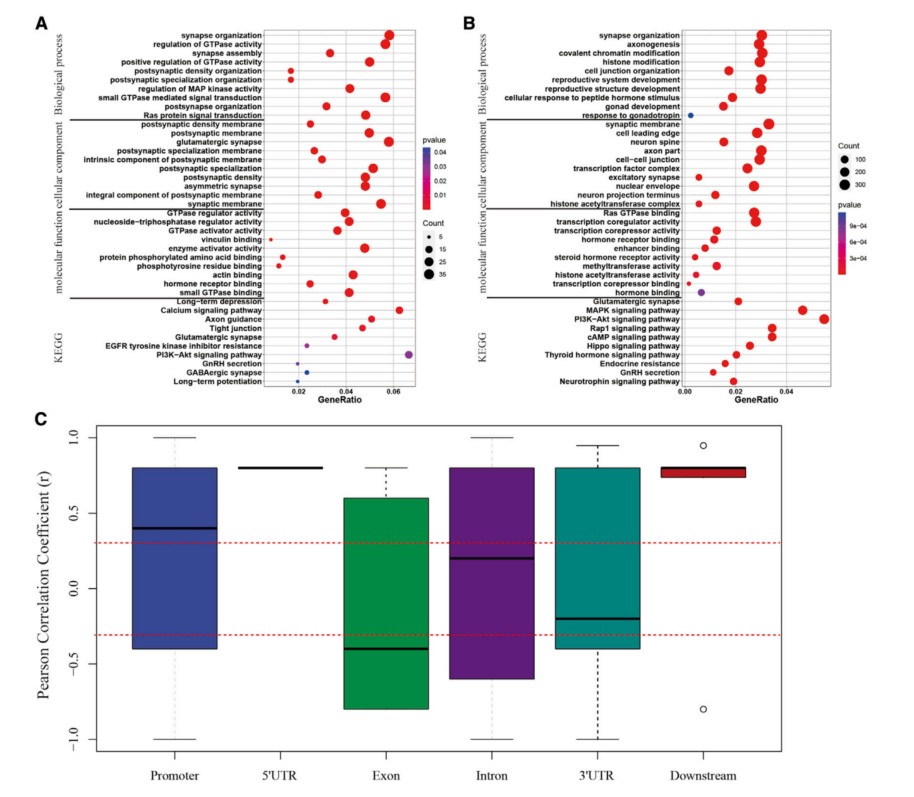
图2 REs的DNA羟甲基化和DNA甲基化及近端基因的表达
3、Htr6的表达受体外SINEs甲基化调控
本研究发现Htr6显著下调,同时在其启动子区域存在一个极低甲基化的SINE。为了研究青春期RE的表观遗传学机制,研究了小鼠下丘脑中产生促性腺激素释放激素(GnRH)的GT1-7细胞中Htr6的转录,该转录可能受到SINEs的DNA甲基化的调节。首先,用Dnmt1、Dnmt3a和Dnmt3b的siRNA处理GT1-7细胞,Htr6启动子上两个CpG岛的DNA甲基化无显著变化(图3A)。同样,由于Dnmt RNA干扰,Htr6启动子内某个SINE的唯一CpG位点出现了轻微的甲基化变化(图3B),表明该SINE的DNA甲基化可能不受DNMTs的调节。有趣的是,用CRISPR-Cas9从GT1-7(GT1-7SINE-CpG-KO1)细胞的该SINE中删除“CG”位点时,与Dnmt3a和Dnmt3b相比,Dnmt1对启动子上的DNA甲基化贡献更大(图3C), Dnmt1处理水平的降低可以增强GT1-7SINE-CpG-KO1细胞中Htr6的表达(图3D和3E)。这些结果表明,SINE甲基化影响GT1-7细胞中dnmt1介导的启动子甲基化和Htr6的表达。
用10 ug/mL L-色氨酸处理GT1-7细胞4 h,阻断GnRH分泌。Htr6启动子在野生型GT1-7细胞中低甲基化,而在GT1-7SINE-CpG-KO1细胞中仍保持高甲基化(图3F)。色氨酸处理后野生型GT1-7细胞d的Htr6表达量上调,但GT1-7SINE-CpG-KO1细胞中没有上调(图3G和3H)。SINEs的DNA甲基化可能是Dnmt抑制Htr6启动子DNA甲基化的独立作用机制。
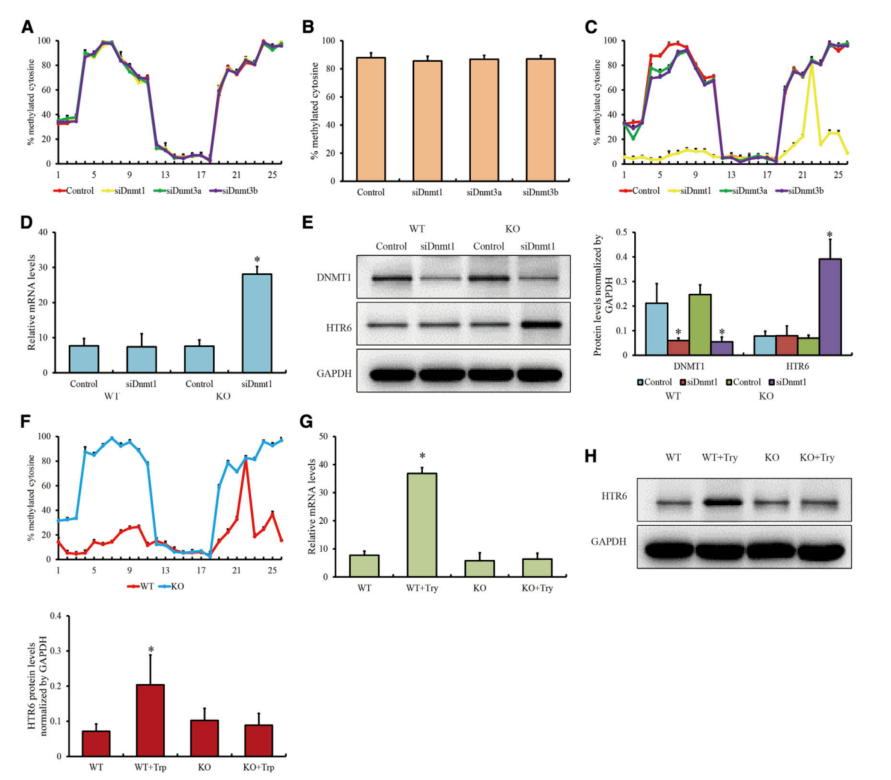
图3 Htr6启动子中SINEs的DNA甲基化和Htr6的表达
4、甲基化的SINE通过NR5A2阻滞Htr6启动子上Dnmt1的富集
分析了DNA甲基化与邻近基因转录正相关的83个REs的一致DNA基序。根据SINE序列(图4A),通过搜索JASPAR数据库,认为核受体NR5A2是顶部结合蛋白。通过ChIP-qPCR检测NR5A2和DNMT1在Htr6启动子上的富集情况,发现野生型GT1-7细胞中NR5A2在Htr6启动子中大量富集,而在GT1-7SINE-CpG-KO1细胞中完全缺失,DNMT1占用的变化与NR5A2相反(图4B)。此外,色氨酸在野生型GT1-7细胞中可增强NR5A2的富集,降低DNMT1对Htr6启动子的亲和力,而在GT1-7SINE-CpG-KO1细胞中无此作用。这些结果表明,甲基化的SINEs可以被NR5A2识别,并抑制Htr6启动子上DNMT1的富集。
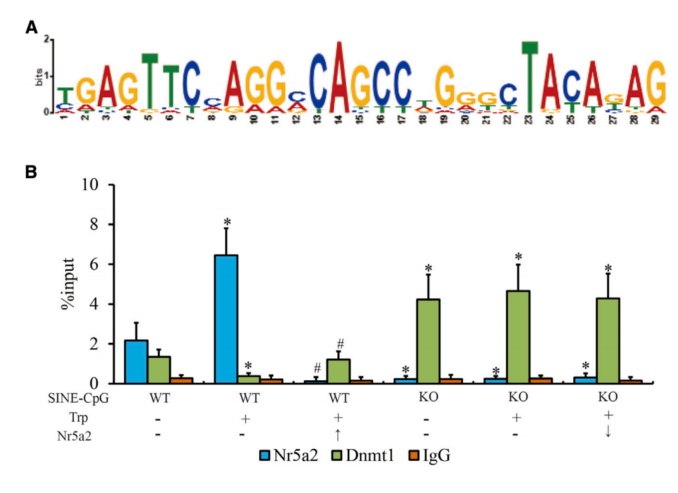
图4 NR5A2对Htr6启动子SINE甲基化的影响
5、SINEs的5hmC对青春期成熟期间ARC增强子和基因表达的影响
PRDM10是维持细胞分化和个体发生过程中全局翻译的重要转录因子。MAP1B对突触成熟至关重要,包括树突棘和轴突的发育。对于DHMREs, 10,685个增强子包含38,405个REs(5302个LINEs,6802个LTRs, 2812个低复杂度序列,114个卫星序列,6531个简单重复序列和16844个SINEs)显示出显著的羟甲基化差异(图5A)。特别是,在8周和4周ARC中SINEs中,5hmC的变化与最接近的基因表达呈正相关(r = 0.615, p = 0.016)(图5B)。
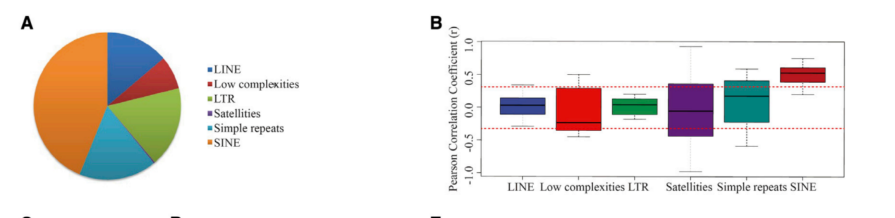
图5 增强子内REs的DNA羟甲基化和DNA甲基化及其靶基因的表达
6、染色质可及性与增强子中羟甲基化SINEs的相关性
为了进一步研究增强子中羟甲基化REs在基因转录表观遗传调控中的作用,用ATAC-seq研究青春期染色质可及性的变化。在4周和8周的ARC中,大多数峰位于远端基因间区(图5C和5D),并确定SINEs的染色质可及性的显著变化与5hmC的变化呈正相关(r = 0.638,p = 0.009)(图5E)。换句话说,REs的DNA羟甲基化的增加通常伴随着增强子区域的染色质开放,如Kiss1、Agmat、Slitrk1和Srp9(图5F)。
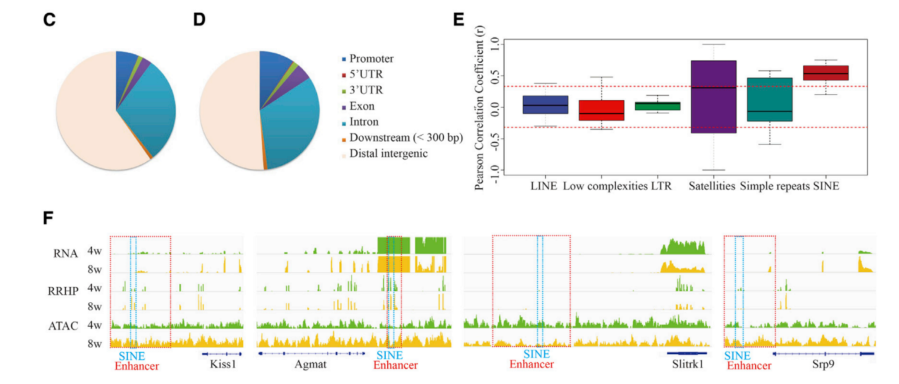
图5 增强子内REs的DNA羟甲基化和DNA甲基化及其靶基因的表达
7、GT1-7细胞上游增强子SINEs的羟甲基化激活Kiss1转录
为了验证DNA羟甲基化对增强子转录活性的影响,研究了两个在Kiss1周围的远端增强子(chr1: 133,308,985-133,319,710;chr1:133,339,139-133,339,571)。巧合的是,Kiss1的上游增强子也包含两个SINEs,其中第一个在8周龄时羟甲基化显著高于4周龄时的ARCs。通过CRISPR-Cas9构建了GT1-7SINE-CpG-KO2细胞,在第一个SINEs上有两个CpG缺失。与野生型细胞相比,雌二醇诱导后GT1-7SINE-CpG-KO2细胞中Kiss1的表达略有下调(图6A和6B)。测定这两种CpGs的DNA甲基化和羟甲基化水平,表明这两种CpGs始终稳定地甲基化,而在雌激素处理的野生型GT1-7细胞中,它们基本上是超羟甲基化的(图6C),表明在该系统中只有5hmC可能归因于增强子活性。
DNase I-qPCR检测结果表明,在GT1-7SINE-CpG-KO2细胞中,整个增强子区域和Kiss1启动子的染色质可及性受到损害(图6D)。同样,ChIP-qPCR检测结果还显示,H3K27ac和雌激素受体α (ESR1)乙酰化水平降低,增强子区H3K27me3甲基化水平升高,表明SINEs羟甲基化的缺失阻滞了ESR1的募集,导致GT1-7SINE-CpG-KO2细胞中引物增强子和Kiss1转录沉默(图6E)。数据表明,羟甲基化的SINEs可能促进Kiss1中增强子的转录激活。

图6 Kiss1表达增强子中SINEs的DNA甲基化
三、研究结论
本研究利用CRISPR-Cas9技术,利用GT1-7细胞建立Kiss1r附近SINEs缺失的体外模型,检测DNA甲基转移酶( DNMT)和TET敲除后Kiss1r的转录。GT1-7细胞上游增强子SINEs的羟甲基化激活Kiss1转录,通常伴随着增强子区域的染色质开放。
参考文献:
The roles of DNA methylation and hydroxymethylation at short interspersed nuclear elements in the hypothalamic ARCuate nucleus during puberty.[J]Molecular Therapy-Nucleic Acids, 2021.